En esta publicación, demostramos cómo ajustar de manera eficiente un modelo de lenguaje de proteínas (pLM) de última generación para predecir la localización subcelular de proteínas utilizando Amazon SageMaker.
Las proteínas son las máquinas moleculares del cuerpo, responsables de todo, desde mover los músculos hasta responder a las infecciones. A pesar de esta variedad, todas las proteínas están formadas por cadenas repetidas de moléculas llamadas aminoácidos. El genoma humano codifica 20 aminoácidos estándar, cada uno con una estructura química ligeramente diferente. Estos pueden representarse mediante letras del alfabeto, lo que luego nos permite analizar y explorar proteínas como una cadena de texto. La enorme cantidad posible de secuencias y estructuras de proteínas es lo que les da a las proteínas su amplia variedad de usos.
Las proteínas también desempeñan un papel clave en el desarrollo de fármacos, como objetivos potenciales pero también como terapias. Como se muestra en la siguiente tabla, muchos de los medicamentos más vendidos en 2022 fueron proteínas (especialmente anticuerpos) u otras moléculas como el ARNm traducido a proteínas en el cuerpo. Debido a esto, muchos investigadores de ciencias biológicas necesitan responder preguntas sobre proteínas de manera más rápida, económica y precisa.
| Nombre | Fabricante | Ventas globales en 2022 (miles de millones de dólares) | Indicaciones |
| Comirnaty | Pfizer/BioNTech | $40.8 | COVID-19 |
| Spikevax | Moderno | $21.8 | COVID-19 |
| Humira | Abbvie | $21.6 | Artritis, enfermedad de Crohn y otras. |
| Keytruda | merck | $21.0 | Varios cánceres |
Fuente de datos: Urquhart, L. Principales empresas y medicamentos por ventas en 2022. Nature Reviews Drug Discovery 22, 260–260 (2023).
Como podemos representar proteínas como secuencias de caracteres, podemos analizarlas utilizando técnicas desarrolladas originalmente para el lenguaje escrito. Esto incluye grandes modelos de lenguaje (LLM) previamente entrenados en enormes conjuntos de datos, que luego pueden adaptarse para tareas específicas, como resúmenes de texto o chatbots. De manera similar, los pLM se entrenan previamente en grandes bases de datos de secuencias de proteínas mediante un aprendizaje autosupervisado y sin etiquetar. Podemos adaptarlos para predecir cosas como la estructura 3D de una proteína o cómo puede interactuar con otras moléculas. Los investigadores incluso han utilizado pLM para diseñar nuevas proteínas desde cero. Estas herramientas no reemplazan la experiencia científica humana, pero tienen el potencial de acelerar el desarrollo preclínico y el diseño de ensayos.
Un desafío con estos modelos es su tamaño. Tanto los LLM como los pLM han crecido en órdenes de magnitud en los últimos años, como se ilustra en la siguiente figura. Esto significa que puede llevar mucho tiempo entrenarlos con suficiente precisión. También significa que es necesario utilizar hardware, especialmente GPU, con grandes cantidades de memoria para almacenar los parámetros del modelo.
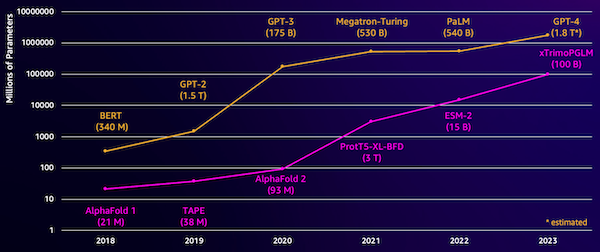
Los tiempos de capacitación prolongados, además de instancias grandes, equivalen a un costo elevado, lo que puede hacer que este trabajo esté fuera del alcance de muchos investigadores. Por ejemplo, en 2023, un equipo de investigación describió el entrenamiento de un pLM de 100 mil millones de parámetros en 768 GPU A100 durante 164 días. Afortunadamente, en muchos casos podemos ahorrar tiempo y recursos adaptando un pLM existente a nuestra tarea específica. Esta técnica se llama sintonia FINAy también nos permite tomar prestadas herramientas avanzadas de otros tipos de modelado de lenguaje.
Descripción general de la solución
El problema específico que abordamos en esta publicación es localización subcelular: Dada una secuencia de proteínas, ¿podemos construir un modelo que pueda predecir si vive en el exterior (membrana celular) o dentro de una célula? Esta es una información importante que puede ayudarnos a comprender la función y si sería un buen objetivo farmacológico.
Comenzamos descargando un conjunto de datos públicos utilizando Amazon SageMaker Studio. Luego usamos SageMaker para ajustar el modelo de lenguaje de proteínas ESM-2 utilizando un método de entrenamiento eficiente. Finalmente, implementamos el modelo como un criterio de valoración de inferencia en tiempo real y lo utilizamos para probar algunas proteínas conocidas. El siguiente diagrama ilustra este flujo de trabajo.
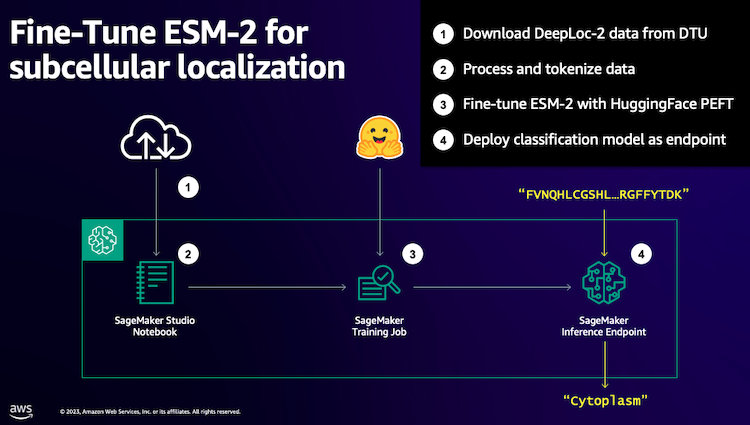
En las siguientes secciones, repasamos los pasos para preparar sus datos de capacitación, crear un script de capacitación y ejecutar un trabajo de capacitación de SageMaker. Todo el código que aparece en esta publicación está disponible en ai-ml-sample-notebooks/tree/main/workshops/Protein_Language_Modelling/finetune_esm_on_deeploc” target=”_blank” rel=”noopener”>GitHub.
Preparar los datos de entrenamiento.
Usamos parte del Conjunto de datos DeepLoc-2, que contiene varios miles de proteínas SwissProt con ubicaciones determinadas experimentalmente. Filtramos secuencias de alta calidad entre 100 y 512 aminoácidos:
df = pd.read_csv(
"https://services.healthtech.dtu.dk/services/DeepLoc-2.0/data/Swissprot_Train_Validation_dataset.csv"
).drop(("Unnamed: 0", "Partition"), axis=1)
df("Membrane") = df("Membrane").astype("int32")
# filter for sequences between 100 and 512 amino acides
df = df(df("Sequence").apply(lambda x: len(x)).between(100, 512))
# Remove unnecessary features
df = df(("Sequence", "Kingdom", "Membrane"))
A continuación, tokenizamos las secuencias y las dividimos en conjuntos de entrenamiento y evaluación:
dataset = Dataset.from_pandas(df).train_test_split(test_size=0.2, shuffle=True)
tokenizer = AutoTokenizer.from_pretrained("facebook/esm2_t33_650M_UR50D")
def preprocess_data(examples, max_length=512):
text = examples("Sequence")
encoding = tokenizer(text, truncation=True, max_length=max_length)
encoding("labels") = examples("Membrane")
return encoding
encoded_dataset = dataset.map(
preprocess_data,
batched=True,
num_proc=os.cpu_count(),
remove_columns=dataset("train").column_names,
)
encoded_dataset.set_format("torch")
Finalmente, cargamos los datos de capacitación y evaluación procesados en Amazon Simple Storage Service (Amazon S3):
train_s3_uri = S3_PATH + "/data/train"
test_s3_uri = S3_PATH + "/data/test"
encoded_dataset("train").save_to_disk(train_s3_uri)
encoded_dataset("test").save_to_disk(test_s3_uri)Crear un guión de entrenamiento
Modo de secuencia de comandos de SageMaker le permite ejecutar su código de capacitación personalizado en contenedores de marco de aprendizaje automático (ML) optimizados administrados por AWS. Para este ejemplo, adaptamos un script existente para clasificación de texto de Abrazando la cara. Esto nos permite probar varios métodos para mejorar la eficiencia de nuestro trabajo de formación.
Método 1: clase de entrenamiento con pesas
Como muchos conjuntos de datos biológicos, los datos de DeepLoc están distribuidos de manera desigual, lo que significa que no hay una cantidad igual de proteínas de membrana y no membrana. Podríamos volver a muestrear nuestros datos y descartar registros de la clase mayoritaria. Sin embargo, esto reduciría los datos totales de entrenamiento y potencialmente perjudicaría nuestra precisión. En cambio, calculamos los pesos de la clase durante el trabajo de entrenamiento y los usamos para ajustar la pérdida.
En nuestro guión de entrenamiento, subclasificamos el Trainer clase de transformers con un WeightedTrainer clase que tiene en cuenta los pesos de clase al calcular la pérdida de entropía cruzada. Esto ayuda a evitar sesgos en nuestro modelo:
class WeightedTrainer(Trainer):
def __init__(self, class_weights, *args, **kwargs):
self.class_weights = class_weights
super().__init__(*args, **kwargs)
def compute_loss(self, model, inputs, return_outputs=False):
labels = inputs.pop("labels")
outputs = model(**inputs)
logits = outputs.get("logits")
loss_fct = torch.nn.CrossEntropyLoss(
weight=torch.tensor(self.class_weights, device=model.device)
)
loss = loss_fct(logits.view(-1, self.model.config.num_labels), labels.view(-1))
return (loss, outputs) if return_outputs else lossMétodo 2: acumulación de gradiente
La acumulación de gradientes es una técnica de entrenamiento que permite a los modelos simular el entrenamiento en lotes de mayor tamaño. Normalmente, el tamaño del lote (la cantidad de muestras utilizadas para calcular el gradiente en un paso de entrenamiento) está limitado por la capacidad de memoria de la GPU. Con la acumulación de gradiente, el modelo calcula primero los gradientes en lotes más pequeños. Luego, en lugar de actualizar los pesos del modelo de inmediato, los gradientes se acumulan en varios lotes pequeños. Cuando los gradientes acumulados igualan el tamaño de lote mayor objetivo, se realiza el paso de optimización para actualizar el modelo. Esto permite que los modelos se entrenen con lotes efectivamente más grandes sin exceder el límite de memoria de la GPU.
Sin embargo, se necesita un cálculo adicional para los pases hacia adelante y hacia atrás de lotes más pequeños. El aumento del tamaño de los lotes mediante la acumulación de gradientes puede ralentizar el entrenamiento, especialmente si se utilizan demasiados pasos de acumulación. El objetivo es maximizar el uso de la GPU pero evitar ralentizaciones excesivas debido a demasiados pasos de cálculo de gradiente adicionales.
Método 3: puntos de control de gradiente
El punto de control de gradiente es una técnica que reduce la memoria necesaria durante el entrenamiento y al mismo tiempo mantiene el tiempo de cálculo razonable. Las redes neuronales grandes consumen mucha memoria porque tienen que almacenar todos los valores intermedios del paso hacia adelante para poder calcular los gradientes durante el paso hacia atrás. Esto puede causar problemas de memoria. Una solución es no almacenar estos valores intermedios, pero luego hay que volver a calcularlos durante el paso hacia atrás, lo que lleva mucho tiempo.
Los puntos de control de gradiente proporcionan un enfoque equilibrado. Guarda sólo algunos de los valores intermedios, llamados puntos de controly recalcula los demás según sea necesario. Por tanto, utiliza menos memoria que almacenarlo todo, pero también menos cálculo que recalcularlo todo. Al seleccionar estratégicamente qué activaciones controlar, el control de gradiente permite entrenar grandes redes neuronales con un uso de memoria y un tiempo de cálculo manejables. Esta importante técnica hace posible entrenar modelos muy grandes que de otro modo tendrían limitaciones de memoria.
En nuestro script de entrenamiento, activamos la activación de gradiente y los puntos de control agregando los parámetros necesarios al TrainingArguments objeto:
from transformers import TrainingArguments
training_args = TrainingArguments(
gradient_accumulation_steps=4,
gradient_checkpointing=True
)Método 4: Adaptación de bajo rango de los LLM
Los modelos de lenguaje grandes como ESM-2 pueden contener miles de millones de parámetros cuyo entrenamiento y ejecución son costosos. Investigadores Desarrolló un método de entrenamiento llamado Adaptación de bajo rango (LoRA) para hacer que el ajuste de estos enormes modelos sea más eficiente.
La idea clave detrás de LoRA es que al ajustar un modelo para una tarea específica, no es necesario actualizar todos los parámetros originales. En cambio, LoRA agrega nuevas matrices más pequeñas al modelo que transforman las entradas y salidas. Sólo estas matrices más pequeñas se actualizan durante el ajuste fino, que es mucho más rápido y utiliza menos memoria. Los parámetros del modelo original permanecen congelados.
Después de realizar ajustes con LoRA, puede fusionar las pequeñas matrices adaptadas nuevamente en el modelo original. O puede mantenerlos separados si desea ajustar rápidamente el modelo para otras tareas sin olvidar las anteriores. En general, LoRA permite que los LLM se adapten de manera eficiente a nuevas tareas a una fracción del costo habitual.
En nuestro script de entrenamiento, configuramos LoRA usando el PEFT biblioteca de Hugging Face:
from peft import get_peft_model, LoraConfig, TaskType
import torch
from transformers import EsmForSequenceClassification
model = EsmForSequenceClassification.from_pretrained(
“facebook/esm2_t33_650M_UR50D”,
Torch_dtype=torch.bfloat16,
Num_labels=2,
)
peft_config = LoraConfig(
task_type=TaskType.SEQ_CLS,
inference_mode=False,
bias="none",
r=8,
lora_alpha=16,
lora_dropout=0.05,
target_modules=(
"query",
"key",
"value",
"EsmSelfOutput.dense",
"EsmIntermediate.dense",
"EsmOutput.dense",
"EsmContactPredictionHead.regression",
"EsmClassificationHead.dense",
"EsmClassificationHead.out_proj",
)
)
model = get_peft_model(model, peft_config)Enviar un trabajo de formación de SageMaker
Una vez que haya definido su guión de capacitación, puede configurar y enviar un trabajo de capacitación de SageMaker. Primero, especifique los hiperparámetros:
hyperparameters = {
"model_id": "facebook/esm2_t33_650M_UR50D",
"epochs": 1,
"per_device_train_batch_size": 8,
"gradient_accumulation_steps": 4,
"use_gradient_checkpointing": True,
"lora": True,
}A continuación, defina qué métricas capturar de los registros de entrenamiento:
metric_definitions = (
{"Name": "epoch", "Regex": "'epoch': ((0-9.)*)"},
{
"Name": "max_gpu_mem",
"Regex": "Max GPU memory use during training: ((0-9.e-)*) MB",
},
{"Name": "train_loss", "Regex": "'loss': ((0-9.e-)*)"},
{
"Name": "train_samples_per_second",
"Regex": "'train_samples_per_second': ((0-9.e-)*)",
},
{"Name": "eval_loss", "Regex": "'eval_loss': ((0-9.e-)*)"},
{"Name": "eval_accuracy", "Regex": "'eval_accuracy': ((0-9.e-)*)"},
)Finalmente, defina un estimador de Hugging Face y envíelo para entrenamiento en un tipo de instancia ml.g5.2xlarge. Este es un tipo de instancia rentable que está ampliamente disponible en muchas regiones de AWS:
from sagemaker.experiments.run import Run
from sagemaker.huggingface import HuggingFace
from sagemaker.inputs import TrainingInput
hf_estimator = HuggingFace(
base_job_name="esm-2-membrane-ft",
entry_point="lora-train.py",
source_dir="scripts",
instance_type="ml.g5.2xlarge",
instance_count=1,
transformers_version="4.28",
pytorch_version="2.0",
py_version="py310",
output_path=f"{S3_PATH}/output",
role=sagemaker_execution_role,
hyperparameters=hyperparameters,
metric_definitions=metric_definitions,
checkpoint_local_path="/opt/ml/checkpoints",
sagemaker_session=sagemaker_session,
keep_alive_period_in_seconds=3600,
tags=({"Key": "project", "Value": "esm-fine-tuning"}),
)
with Run(
experiment_name=EXPERIMENT_NAME,
sagemaker_session=sagemaker_session,
) as run:
hf_estimator.fit(
{
"train": TrainingInput(s3_data=train_s3_uri),
"test": TrainingInput(s3_data=test_s3_uri),
}
)La siguiente tabla compara los diferentes métodos de entrenamiento que analizamos y su efecto en el tiempo de ejecución, la precisión y los requisitos de memoria GPU de nuestro trabajo.
| Configuración | Tiempo facturable (min) | Precisión de la evaluación | Uso máximo de memoria de GPU (GB) |
| Modelo básico | 28 | 0,91 | 22.6 |
| Base + GA | 21 | 0,90 | 17.8 |
| Base + CG | 29 | 0,91 | 10.2 |
| Base + LORA | 23 | 0,90 | 18.6 |
Todos los métodos produjeron modelos con alta precisión de evaluación. El uso de LoRA y la activación de gradiente redujeron el tiempo de ejecución (y el costo) en un 18% y un 25%, respectivamente. El uso de puntos de control de gradiente redujo el uso máximo de memoria de la GPU en un 55 %. Dependiendo de sus limitaciones (costo, tiempo, hardware), uno de estos enfoques puede tener más sentido que otro.
Cada uno de estos métodos funciona bien por sí solo, pero ¿qué sucede cuando los usamos en combinación? La siguiente tabla resume los resultados.
| Configuración | Tiempo facturable (min) | Precisión de la evaluación | Uso máximo de memoria de GPU (GB) |
| Todos los métodos | 12 | 0,80 | 3.3 |
En este caso, vemos una reducción del 12% en la precisión. Sin embargo, hemos reducido el tiempo de ejecución en un 57 % y el uso de memoria de la GPU en un 85 %. Esta es una disminución masiva que nos permite capacitarnos en una amplia gama de tipos de instancias rentables.
Limpiar
Si está siguiendo su propia cuenta de AWS, elimine los puntos finales de inferencia en tiempo real y los datos que creó para evitar cargos adicionales.
predictor.delete_endpoint()
bucket = boto_session.resource("s3").Bucket(S3_BUCKET)
bucket.objects.filter(Prefix=S3_PREFIX).delete()Conclusión
En esta publicación, demostramos cómo ajustar de manera eficiente modelos de lenguaje de proteínas como ESM-2 para una tarea científicamente relevante. Para obtener más información sobre el uso de las bibliotecas Transformers y PEFT para entrenar pLMS, consulte las publicaciones Aprendizaje profundo con proteínas y ESMBind (ESMB): Adaptación de rango bajo de ESM-2 para la predicción del sitio de unión a proteínas en el blog Hugging Face. También puede encontrar más ejemplos del uso del aprendizaje automático para predecir las propiedades de las proteínas en el Impresionante análisis de proteínas en AWS Repositorio de GitHub.
Sobre el Autor
 Brian Leal es arquitecto senior de soluciones de IA/ML en el equipo global de atención médica y ciencias biológicas de Amazon Web Services. Tiene más de 17 años de experiencia en biotecnología y aprendizaje automático, y le apasiona ayudar a los clientes a resolver desafíos genómicos y proteómicos. En su tiempo libre le gusta cocinar y comer con sus amigos y familiares.
Brian Leal es arquitecto senior de soluciones de IA/ML en el equipo global de atención médica y ciencias biológicas de Amazon Web Services. Tiene más de 17 años de experiencia en biotecnología y aprendizaje automático, y le apasiona ayudar a los clientes a resolver desafíos genómicos y proteómicos. En su tiempo libre le gusta cocinar y comer con sus amigos y familiares.
 NEWSLETTER
NEWSLETTER





