La patología digital es esencial para el diagnóstico y el tratamiento del cáncer, jugando un papel fundamental en la prestación de salud y el desarrollo farmacéutico y el desarrollo. La patología tradicionalmente depende en gran medida de la experiencia y la experiencia del patólogo para realizar un examen meticuloso de muestras de tejidos para identificar anormalidades. Sin embargo, el aumento de la complejidad y el volumen de casos requieren herramientas avanzadas para ayudar a los patólogos a hacer diagnósticos más rápidos y precisos.
La digitalización de las diapositivas de patología, conocidas como imágenes de diapositivas completas (WSI), dio lugar al nuevo campo de la patología computacional. Al aplicar ai a estos WSI digitalizados, los investigadores están trabajando para desbloquear nuevas ideas y mejorar los flujos de trabajo de anotaciones actuales. Un avance fundamental en el campo de la patología computacional ha sido el surgimiento de arquitecturas de redes neuronales profundas a gran escala, conocidas como modelos de fundación (FMS). Estos modelos se entrenan utilizando algoritmos de aprendizaje auto-supervisados en conjuntos de datos expansivos, lo que les permite capturar un repertorio integral de representaciones visuales y patrones inherentes a las imágenes de patología. El poder de los FMS radica en su capacidad para aprender incrustaciones de datos robustas y generalizables que pueden transferirse y ajustar de manera efectiva para una amplia variedad de tareas aguas abajo, desde la detección automatizada de la enfermedad y la caracterización del tejido hasta el análisis cuantitativo de biomarcadores y la subtasa patológica.
Recientemente, startup francesa Bioptimus anunció el <a target="_blank" href="http://bioptimus.com/news/bioptimus-launches-h-optimus-0-the-worlds-largest-open-source-ai-foundation-model-for-pathology” target=”_blank” rel=”noopener”>liberar de una nueva visión de patología FM: H-Optimus-0, el FM públicamente más grande del mundo para la patología. Con 1.100 millones de parámetros, H-Optimus-0 fue entrenado en un conjunto de datos patentado de varios cientos de millones de imágenes extraídas de más de 500,000 diapositivas de histopatología. Esto establece un nuevo punto de referencia para el rendimiento de vanguardia en tareas de diagnóstico médico crítico, desde identificar células cancerosas hasta detectar anormalidades genéticas en los tumores.
La reciente incorporación de H-Optimus-0 a amazon Sagemaker Jumpstart marca un hito significativo para hacer que las capacidades de IA avanzadas sean accesibles para las organizaciones de atención médica. Este poderoso FM, con su capacitación integral en más de 500,000 diapositivas de histopatología, representa una herramienta valiosa para las organizaciones que buscan mejorar sus flujos de trabajo de patología digital.
En esta publicación, demostramos cómo usar H-Optimus-0 para dos tareas de patología digital comunes: análisis a nivel de parche para un examen detallado de tejido y análisis a nivel de deslizamiento para una evaluación de diagnóstico más amplia. A través de ejemplos prácticos, le mostramos cómo adaptar este FM a estos casos de uso específicos al tiempo que optimizamos los recursos computacionales.
Descripción general de la solución
Nuestra solución utiliza el ecosistema integrado AWS para crear una tubería escalable eficiente para los flujos de trabajo de IA de patología digital. La arquitectura combina los siguientes servicios:
El siguiente diagrama ilustra la arquitectura de soluciones para capacitación y la implementación de FMS ajustados utilizando H-Optimus-0.
Este diagrama ilustra la arquitectura de soluciones para capacitar e implementar FMS ajustados utilizando H-Optimus-0
Esta publicación proporciona scripts de ejemplo y cuadernos de capacitación a continuación. <a target="_blank" href="https://github.com/aws-samples/ai-digital-pathology/tree/main” target=”_blank” rel=”noopener”>Repositorio de Github.
Requisitos previos
Asumimos que tiene acceso y se autentica en una cuenta de AWS. La plantilla de AWS CloudFormation para esta solución utiliza instancias T3.Medium para alojar el cuaderno de Sagemaker. La extracción de características utiliza tipos de instancia G5.2xLarge impulsados por NVIDIA T4 GPU probado en la región de AWS de EE. UU.-Oeste-2. Los trabajos de capacitación se ejecutan en instancias P3.2xLarge y G5.2xLarge. Consulte sus cuotas de servicio AWS para asegurarse de tener suficiente acceso a estos tipos de instancias.
Crea la infraestructura de AWS
Para comenzar con los flujos de trabajo de IA de patología, utilizamos AWS CloudFormation para automatizar la configuración de nuestra infraestructura central. El proporcionado <a target="_blank" href="https://github.com/aws-samples/ai-digital-pathology/blob/main/infra/infra-stack.yml” target=”_blank” rel=”noopener”>infra-stack.yml La plantilla crea un entorno completo listo para el modelo y la capacitación.
Nuestra pila CloudFormation configura un entorno de red seguro utilizando amazon Virtual Private Cloud (amazon VPC), estableciendo subredes públicas y privadas con puertas de enlace apropiadas para la conectividad a Internet. Dentro de esta red, crea un sistema de archivos EFS para almacenar y servir de manera eficiente imágenes de diapositivas de gran patología. La pila también disposición una instancia de cuaderno de Sagemaker que se conecta automáticamente al almacenamiento EFS, proporcionando un acceso perfecto a los datos de capacitación.
La plantilla maneja todas las configuraciones de seguridad necesarias, incluidas las roles de AWS Identity and Access Management (IAM). Al implementar la pila, tome nota de los identificadores privados de la subred y el grupo de seguridad; Deberá asegurarse de que sus trabajos de capacitación puedan acceder al almacenamiento de datos EFS.
Para obtener instrucciones de configuración y opciones de configuración detalladas, consulte el ReadMe en nuestro <a target="_blank" href="https://github.com/aws-samples/ai-digital-pathology/tree/main” target=”_blank” rel=”noopener”>Repositorio de Github.
Use FMS para tareas de predicción a nivel de parche
El análisis de nivel de parche es fundamental para los flujos de trabajo de IA de patología digital. En lugar de procesar WSIS enteros que pueden exceder varios gigabytes, el análisis a nivel de parche se centra en regiones de tejido específicas. Este enfoque dirigido permite una utilización eficiente de recursos y ciclos de desarrollo de modelos más rápidos. El siguiente diagrama ilustra el flujo de trabajo de las tareas de predicción de nivel de parche en un WSI.
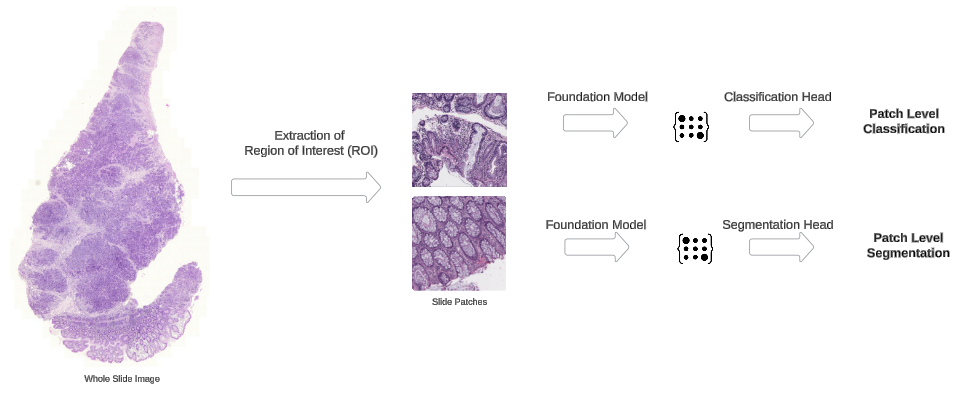
Este diagrama ilustra el flujo de trabajo de las tareas de predicción de nivel de parche en un WSI
Tarea de clasificación: conjunto de datos MHIST
Demostramos la clasificación de nivel de parche utilizando el conjunto de datos MHIST, que contiene imágenes de pólipo colorrectal. La detección temprana de pólipos potencialmente cancerosos afecta directamente las tasas de supervivencia del paciente, lo que hace que este sea un caso de uso clínicamente relevante. Al agregar un cabezal de clasificación simple además de las características previas al estado previo de H-Optimus-0 y al usar sondeo lineal, logramos una precisión del 83%. La implementación utiliza amazon EFS para una transmisión de datos eficiente y instancias P3.2xLarge para una utilización óptima de GPU.
Para acceder al conjunto de datos MHIST, envíe una solicitud de datos a través de su portal para obtener el archivo annotations.csv e imágenes.zip. Nuestro repositorio incluye un script download_mhist.sh que descarga y organiza automáticamente los datos en su almacenamiento EFS.
Tarea de segmentación: conjunto de datos lagarto
Para nuestra segunda tarea a nivel de parche, demostramos segmentación nuclear utilizando el conjunto de datos lagarto, que requiere predicciones precisas a nivel de píxel de los límites nucleares en el tejido del colon. Adaptamos H-Optimus-0 para la segmentación agregando un cabezal adaptador de VIT Mask2Former, lo que permite que el modelo genere máscaras de segmentación detalladas mientras usa las potentes capacidades de extracción de características del FM.
El conjunto de datos de lagarto está disponible en Kaggley nuestro repositorio incluye scripts para descargar automáticamente y preparar los datos para la capacitación. La implementación de segmentación se ejecuta en instancias G5.16xLarge para manejar las demandas computacionales de las predicciones a nivel de píxel.
Use FMS para tareas de nivel de WSI
El análisis de WSIS entero presenta desafíos únicos debido a su tamaño masivo, a menudo superior a 50,000 x 50,000 píxeles. Para abordar esto, implementamos el aprendizaje de instancias múltiples (MIL), que trata cada WSI como una colección de parches más pequeños. Nuestro enfoque MIL basado en la atención aprende automáticamente qué regiones son más relevantes para la predicción final. El siguiente diagrama ilustra el flujo de trabajo para las tareas de predicción de nivel WSI usando MIL.
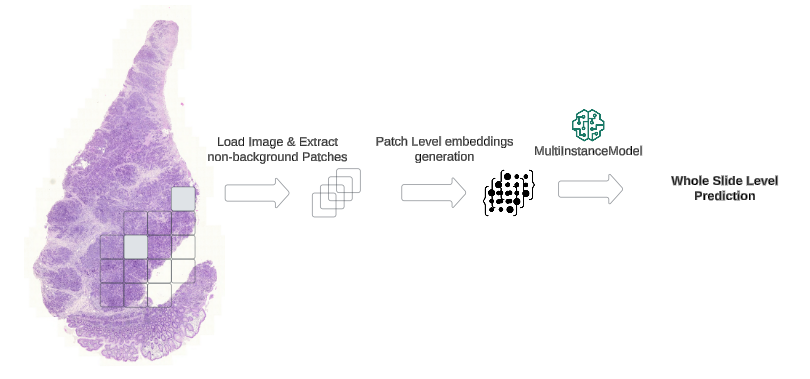
Este diagrama ilustra el flujo de trabajo para las tareas de predicción de nivel WSI usando MIL
Tubería de procesamiento de WSI
Nuestra implementación optimiza el análisis WSI a través de los siguientes métodos:
- Parcheo inteligente: usamos la GPU acelerada <a target="_blank" href="https://docs.rapids.ai/api/cucim/stable/” target=”_blank” rel=”noopener”>Biblioteca de cucim Para cargar de manera eficiente WSIS y aplicar la detección de bordes canny para identificar y extraer solo regiones que contienen tejidos
- Extracción de características: los parches seleccionados se procesan en paralelo utilizando la aceleración de la GPU, con características almacenadas en formato HDF5 eficiente en el espacio para el análisis posterior
Predicción de estado de MSI
Demostramos nuestra tubería WSI mediante la predicción del estado de inestabilidad de microsatélites (MSI), un biomarcador crucial que guía las decisiones de inmunoterapia en el tratamiento del cáncer. El Conjunto de datos TCGA-Coad Se utiliza para esta tarea a través de la Portal de datos GDCy nuestro <a target="_blank" href="https://github.com/aws-samples/ai-digital-pathology/blob/main/README.md” target=”_blank” rel=”noopener”>repositorio Proporciona instrucciones detalladas para descargar el WSIS y correspondiente Etiquetas de MSI.
Limpiar
Después de que haya terminado, no olvide eliminar los recursos asociados (amazon EFS Storage y las instancias de cuaderno de Sagemaker) para evitar costos inesperados.
Conclusión
En esta publicación, demostramos cómo puede usar los servicios de AWS para construir flujos de trabajo de IA digital de patología escalable utilizando el H-Optimus-0 FM. A través de ejemplos prácticos de ambas tareas a nivel de parche (clasificación MHIST y segmentación nuclear de lagarto) y análisis WSI (predicción del estado de MSI), mostramos cómo manejar eficientemente los desafíos únicos de la patología computacional.
Nuestra implementación destaca la integración perfecta entre los servicios de AWS para manejar el procesamiento de datos de patología a gran escala. Aunque utilizamos amazon EFS para esta demostración para permitir flujos de trabajo de capacitación de alto rendimiento, las implementaciones de producción pueden considerar la salud de AWS para el almacenamiento a largo plazo de datos de imágenes médicas.
Esperamos que esta tubería sirva como punto de partida para sus propias iniciativas de patología de IA. El proporcionado <a target="_blank" href="https://github.com/aws-samples/ai-digital-pathology/tree/main” target=”_blank” rel=”noopener”>Repositorio de Github Contiene los componentes necesarios para ayudarlo a comenzar a construir y escalar flujos de trabajo de patología para sus casos de uso específicos. Puede clonar el repositorio y configurar la infraestructura utilizando la plantilla de CloudFormation proporcionada. Luego, intente ajustar H-Optimus-0 en sus propios conjuntos de datos de patología y tareas aguas abajo y compare los resultados con sus métodos actuales.
Nos encantaría saber sobre sus experiencias e ideas. Comuníquese con nosotros o contribuya al FMS disponible públicamente para ayudar a avanzar en el campo de la patología computacional.
Sobre los autores
<img loading="lazy" class="wp-image-97859 size-full alignleft" src="https://technicalterrence.com/wp-content/uploads/2025/02/1738451489_964_Acelerar-los-flujos-de-trabajo-de-anotacion-de-diapositivas-de.png" alt="Pierre de Malliard es un arquitecto senior de soluciones de IA/ML en amazon Web Services y apoya a los clientes en la industria de la salud y las ciencias de la vida. ” width=”100″ height=”109″/>Malliard Pierre es un arquitecto senior de soluciones de IA/ML en amazon Web Services y apoya a los clientes en la industria de la salud y las ciencias de la vida. En su tiempo libre, a Pierre le gusta esquiar y explorar la escena alimentaria de Nueva York.
 Christopher es un gerente de cuentas asociado senior en amazon Web Services (AWS), que ayuda a los proveedores de software independientes (ISVS) innovar, construir y vender las soluciones de software de salud como servicio (SAAS) basado en la nube en el sector público. Parte de la Comunidad de Campo Técnico de Ciencias de la Salud y Ciencias de la Vida (TFC), Christopher tiene como objetivo acelerar la digitalización y utilización de datos de atención médica para impulsar mejores resultados y prestación de atención personalizada.
Christopher es un gerente de cuentas asociado senior en amazon Web Services (AWS), que ayuda a los proveedores de software independientes (ISVS) innovar, construir y vender las soluciones de software de salud como servicio (SAAS) basado en la nube en el sector público. Parte de la Comunidad de Campo Técnico de Ciencias de la Salud y Ciencias de la Vida (TFC), Christopher tiene como objetivo acelerar la digitalización y utilización de datos de atención médica para impulsar mejores resultados y prestación de atención personalizada.
 NEWSLETTER
NEWSLETTER




