A pesar de los avances recientes en el campo de la inteligencia artificial medica (AI), la mayoría de los modelos existentes son angosto, tarea única sistemas que requieren grandes cantidades de datos etiquetados para entrenar. Además, estos modelos no se pueden reutilizar fácilmente en nuevos contextos clínicos, ya que a menudo requieren la recopilación, desidentificación y anotación de datos específicos del sitio para cada nuevo entorno de implementación, que es a la vez laborioso y costoso. este problema de generalización eficiente de datos (la capacidad de un modelo para generalizar a nuevos entornos utilizando un mínimo de datos nuevos) sigue siendo un desafío traslacional clave para los modelos médicos de aprendizaje automático (ML) y, a su vez, ha impedido su amplia aceptación en entornos de atención médica del mundo real.
El surgimiento de modelos de cimientos ofrece una oportunidad importante para repensar el desarrollo de la IA médica para que sea más eficiente, segura y equitativa. Estos modelos se entrenan utilizando datos a escala, a menudo mediante aprendizaje autosupervisado. Este proceso da como resultado modelos generalistas que se pueden adaptar rápidamente a nuevas tareas y entornos con menos necesidad de datos supervisados. Con los modelos básicos, puede ser posible implementar modelos de manera segura y eficiente en varios contextos y entornos clínicos.
En “Imágenes médicas sólidas y eficientes con autosupervisión” (REMEDIS), a ser publicado en Naturaleza Ingeniería Biomédica, presentamos un marco de aprendizaje autosupervisado unificado a gran escala para construir modelos básicos de imágenes médicas. Esta estrategia combina gran escala aprendizaje de transferencia supervisado con aprendizaje autosupervisado y requiere una mínima personalización específica de la tarea. REMEDIS muestra una mejora significativa en la generalización eficiente de datos en las tareas y modalidades de imágenes médicas con una reducción de 3 a 100 veces en los datos específicos del sitio para adaptar modelos a nuevos contextos y entornos clínicos. Sobre la base de esto, nos complace anunciar Fundamentos de investigación de IA médica (alojado por FisioNet), una expansión del lanzamiento público de Chest X-ray Foundations en 2022. Medical AI Research Foundations es una colección de modelos no diagnósticos de código abierto (comenzando con los modelos REMEDIS), API y recursos para ayudar a los investigadores y desarrolladores a acelerar la investigación médica. Investigación de IA.
Autosupervisión a gran escala para imágenes médicas
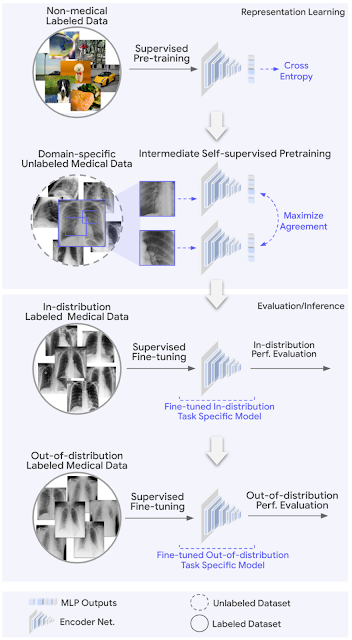
REMEDIS utiliza una combinación de imágenes naturales (no médicas) e imágenes médicas sin etiquetar para desarrollar sólidos modelos básicos de imágenes médicas. Su estrategia de pre-entrenamiento consta de dos pasos. El primero implica el aprendizaje de representación supervisado en un conjunto de datos a gran escala de imágenes naturales etiquetadas (extraídas de Imagen 21k o JFT) utilizando el método Big Transfer (BiT).
El segundo paso implica el aprendizaje autosupervisado intermedio, que no requiere etiquetas y, en cambio, entrena un modelo para aprender representaciones de datos médicos independientemente de las etiquetas. El enfoque específico utilizado para las representaciones previas al entrenamiento y el aprendizaje es SimCLR. El método funciona maximizando el acuerdo entre vistas aumentadas de manera diferente del mismo ejemplo de entrenamiento a través de una pérdida de contraste en una capa oculta de una red neuronal de avance con perceptrón multicapa (MLP) salidas. Sin embargo, REMEDIS es igualmente compatible con otros métodos contrastivos de aprendizaje autosupervisado. Este método de entrenamiento es aplicable para entornos de atención médica, ya que muchos hospitales adquieren datos sin procesar (imágenes) como práctica de rutina. Si bien se tendrían que implementar procesos para que estos datos se puedan utilizar dentro de los modelos (es decir, el consentimiento del paciente antes de recopilar los datos, la desidentificación, etc.), la tarea costosa, lenta y difícil de etiquetar esos datos podría evitarse. utilizando REMEDIS.
Dadas las restricciones de los parámetros del modelo de ML, es importante que nuestro enfoque propuesto funcione cuando se usan tamaños de arquitectura de modelo pequeños y grandes. Para estudiar esto en detalle, consideramos dos arquitecturas ResNet con multiplicadores de profundidad y ancho de uso común, ResNet-50 (1×) y ResNet-152 (2×) como las redes de codificador troncales.
Después del entrenamiento previo, el modelo se ajustó utilizando datos médicos específicos de la tarea etiquetados y se evaluó el desempeño de la tarea en distribución. Además, para evaluar la generalización eficiente de los datos, el modelo también se ajustó opcionalmente utilizando pequeñas cantidades de datos fuera de distribución (OOD).
Evaluación y resultados
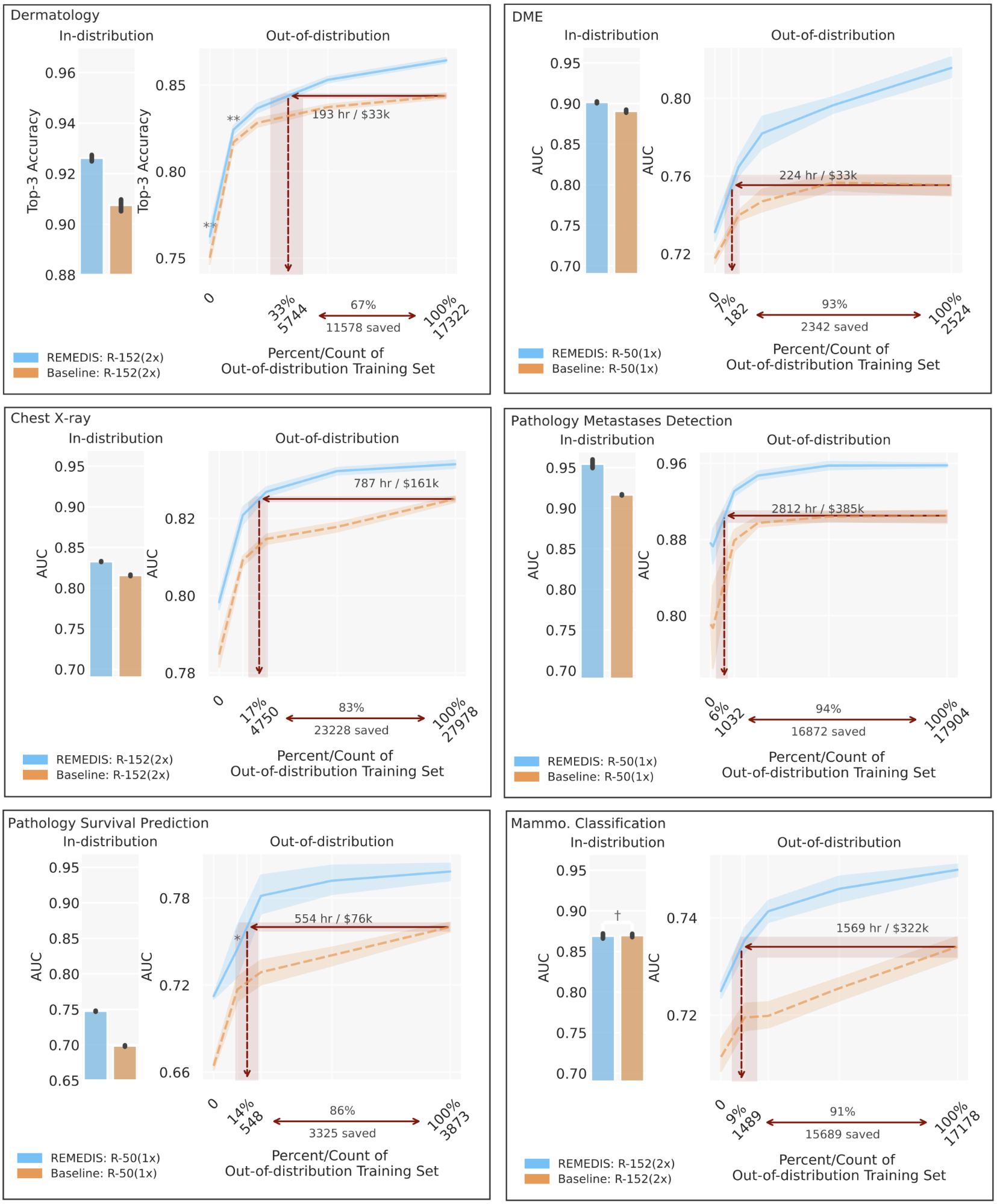
Para evaluar el rendimiento del modelo REMEDIS, simulamos escenarios realistas utilizando datos anonimizados retrospectivos en una amplia gama de tareas y modalidades de imágenes médicas, que incluyen dermatología, imágenes de la retina, interpretación de radiografías de tórax, patología y mamografía. Además, presentamos la noción de generalización eficiente en datos, capturando la capacidad del modelo para generalizar a nuevas distribuciones de implementación con una necesidad significativamente reducida de datos anotados por expertos del nuevo entorno clínico. El rendimiento dentro de la distribución se mide como (1) una mejora en la generalización de tiro cero a la configuración de OOD (evaluación del rendimiento en un conjunto de evaluación de OOD, sin acceso a los datos de entrenamiento del conjunto de datos de OOD) y (2) una reducción significativa en la necesidad de anotación datos de la configuración OOD para alcanzar un rendimiento equivalente al de los expertos clínicos (o umbral que demuestra la utilidad clínica). REMEDIS muestra un rendimiento en distribución significativamente mejorado con una mejora relativa de hasta un 11,5 % en la precisión del diagnóstico con respecto a una línea base fuertemente supervisada.
Lo que es más importante, nuestra estrategia conduce a la generalización eficiente de los datos de los modelos de imágenes médicas, lo que hace coincidir sólidas líneas de base supervisadas que dan como resultado una reducción de 3 a 100 veces en la necesidad de volver a entrenar los datos. Si bien SimCLR es el principal enfoque de aprendizaje autosupervisado utilizado en el estudio, también mostramos que REMEDIS es compatible con otros enfoques, como MoCo-V2, RELIQUIA y Gemelos Barlow. Además, el enfoque funciona en todos los tamaños de arquitectura del modelo.
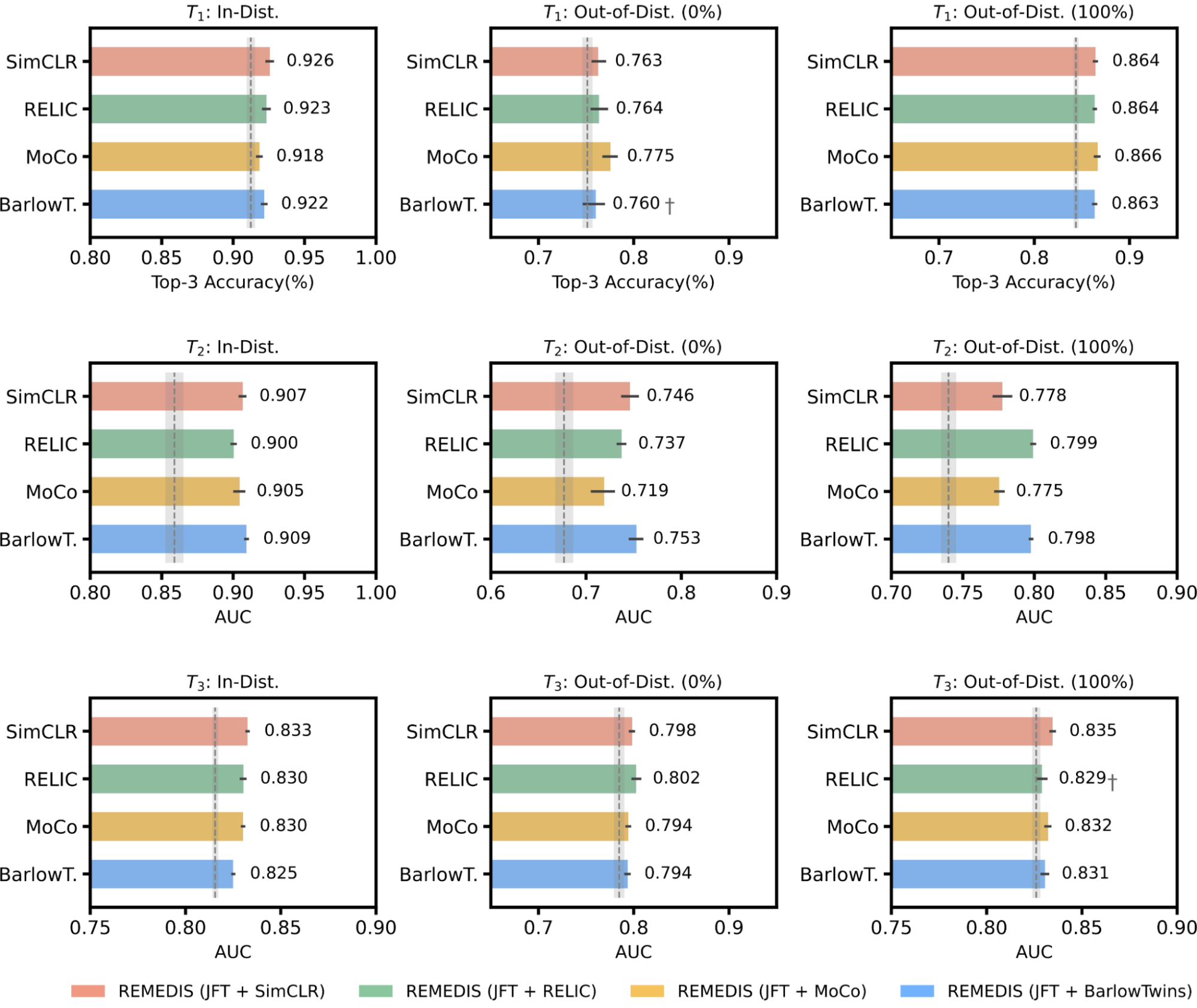 |
| REMEDIS es compatible con MoCo-V2, RELIQUIA y Gemelos Barlow como estrategias alternativas de aprendizaje autosupervisado. Todas las variantes de REMEDIS conducen a mejoras de generalización de datos eficientes sobre la línea de base supervisada fuerte para la clasificación de condiciones dermatológicas (T1), clasificación del edema macular diabético (T2), y clasificación de la condición de la radiografía de tórax (T3). El área sombreada en gris indica el rendimiento de la línea de base supervisada fuerte preentrenada en JFT. |
Fundamentos de investigación de IA médica
Sobre la base de REMEDIS, nos complace anunciar Fundamentos de investigación de IA médica, una expansión del lanzamiento público de Chest X-ray Foundations en 2022. Medical AI Research Foundations es un repositorio de modelos de fundaciones médicas de código abierto alojado por PhysioNet. Esto amplía el enfoque anterior basado en API para abarcar también modelos que no son de diagnóstico, para ayudar a los investigadores y desarrolladores a acelerar su investigación en IA médica. Creemos que REMEDIS y el lanzamiento de Medical AI Research Foundations son un paso hacia la construcción de modelos médicos que pueden generalizarse en entornos y tareas de atención médica.
Estamos sembrando fundaciones de investigación de IA médica con modelos REMEDIS para radiografía de tórax y patología (con información relacionada). código). Mientras que el enfoque de la Fundación de rayos X de tórax existente se centra en proporcionar incrustaciones congeladas para el ajuste fino específico de la aplicación a partir de un modelo entrenado en varios grandes conjuntos de datos privados, los modelos REMEDIS (entrenados en conjuntos de datos públicos) permiten a los usuarios realizar ajustes de extremo a extremo. para su aplicación y para ejecutarse en dispositivos locales. Recomendamos a los usuarios que prueben diferentes enfoques en función de sus necesidades únicas para la aplicación deseada. Esperamos agregar más modelos y recursos para entrenar modelos básicos médicos, como conjuntos de datos y puntos de referencia en el futuro. También damos la bienvenida a la comunidad de investigación de IA médica para que contribuya a esto.
Conclusión
Estos resultados sugieren que REMEDIS tiene el potencial de acelerar significativamente el desarrollo de sistemas ML para imágenes médicas, que pueden preservar su sólido rendimiento cuando se implementan en una variedad de contextos cambiantes. Creemos que este es un importante paso adelante para que la IA de imágenes médicas tenga un amplio impacto. Más allá de los resultados experimentales presentados, el enfoque y los conocimientos descritos aquí se han integrado en varios de Proyectos de investigación de imágenes médicas de Google, como dermatología, mamografía y radiología entre otros. Estamos utilizando un enfoque de aprendizaje autosupervisado similar con nuestros esfuerzos de modelo de base sin imágenes, como Med-Palm y Med-Palma 2.
Con REMEDIS, demostramos el potencial de los modelos básicos para aplicaciones de imágenes médicas. Dichos modelos tienen interesantes posibilidades en aplicaciones médicas con la oportunidad de aprendizaje de representación multimodal. La práctica de la medicina es intrínsecamente multimodal e incorpora información de imágenes, registros de salud electrónicos, sensores, dispositivos portátiles, genómica y más. Creemos que los sistemas de aprendizaje automático que aprovechan estos datos a escala mediante el aprendizaje autosupervisado con una consideración cuidadosa de la privacidad, la seguridad, la equidad y la ética ayudarán a sentar las bases para la próxima generación de sistemas de salud de aprendizaje que escalan la atención médica de clase mundial para todos.
Agradecimientos
Este trabajo involucró extensos esfuerzos de colaboración de un equipo multidisciplinario de investigadores, ingenieros de software, médicos y colaboradores multifuncionales en Google Health AI y Google Brain. En particular, nos gustaría agradecer a nuestro primer coautor, Jan Freyberg, y a nuestros principales autores principales de estos proyectos, Vivek Natarajan, Alan Karthikesalingam, Mohammad Norouzi y Neil Houlsby, por sus valiosas contribuciones y apoyo. También agradecemos a Lauren Winer, Sami Lachgar, Yun Liu y Karan Singhal por sus comentarios sobre esta publicación y a Tom Small por su apoyo en la creación de las imágenes. Finalmente, también agradecemos al equipo de PhysioNet por su apoyo para albergar fundaciones de investigación de IA médica. Los usuarios que tengan preguntas pueden comunicarse con medical-ai-research-foundations en google.com.